摘要:研究通過snm3C-seq結合snRNA-seq,首次繪制了人類皮下脂肪組織(SAT)的細胞類型分辨率表觀基因組圖譜。
多組學解析人類皮下脂肪細胞異質性
研究團隊采用單核甲基化-3C測序(snm3C-seq)技術,對5例芬蘭女性皮下脂肪組織(SAT)活檢樣本進行多組學分析,同步捕獲6,652個核的DNA甲基化(mCG)和染色質構象數據,并與29,423個核的snRNA-seq數據整合。通過5kb分辨率mCG分析和100kb染色質互作聚類,鑒定出脂肪細胞、脂肪干細胞祖細胞(ASPCs)、血管周圍細胞等7種主要細胞類型,并發現63個核呈現表觀遺傳"過渡態"特征——其染色質構象類似脂肪細胞而甲基化模式接近血管周圍細胞,暗示人類脂肪細胞可能通過該路徑分化。

圖1 人類皮下脂肪組織的單細胞 DNA 甲基化組與三維基因組圖譜
甲基化與轉錄組的跨模態調控網絡
基因體mCG低甲基化與高表達呈現強相關性(如脂肪標記基因GPAM)。跨模態分析發現PPAR信號通路關鍵基因(ACSL1、ADIPOQ等)在脂肪細胞中同步呈現低mCG和高表達,而脂肪分化基因(如LEP、SREBF1)則富集于脂肪細胞特異性低甲基化區域。值得注意的是,僅21%的脂肪細胞標記基因在兩種模態中重疊,但63%相關生物學通路(如脂質代謝)具有跨模態一致性,提示表觀遺傳與轉錄調控的協同作用。
髓系與脂肪細胞的甲基化"蹺蹺板"效應
全基因組共鑒定705,063個差異甲基化區域(DMRs),覆蓋5.39%基因組。脂肪細胞與ASPCs分別呈現56.3%和50.6%區域低甲基化,而髓系細胞則73.0%區域高甲基化,47.3% DMRs呈現此消彼長模式。HOMER分析揭示髓系細胞低甲基化區域富集IRF4、MEF2B等轉錄因子結合基序,ENCODE的ChIP-seq數據驗證這些基序在GM12878細胞中富集度達3.5倍(P<1×10-12)。
脂肪細胞特異的基因組三維結構
脂肪細胞表現出獨特的染色質空間特征:短程互作(100kb-2Mb)比例較其他細胞高1.51倍(P<10-91),25kb分辨率下檢測到更多結構域(平均4,120個/細胞)。染色體區室分析顯示,脂肪細胞的A區室(活性染色質)與ADIPOQ等肥胖GWAS變異顯著共定位,而髓系細胞A區室則富集炎癥相關變異。值得注意的是,11,571個差異區室區域中,ADIPOQ基因所在基因組區域在ASPCs中處于B區室(抑制狀態),而在脂肪細胞中轉為A區室。
TET1-DNMT3A的表觀遺傳調控軸
單細胞轉錄組顯示去甲基化酶TET1在脂肪細胞特異性高表達(P<10-300),而甲基轉移酶DNMT3A在髓系細胞優勢表達(P<10-136)。人類前脂肪細胞分化實驗證實,TET1與PPARγ等脂肪生成基因呈現同步表達波動。SAT批量RNA-seq進一步發現,TET1表達與胰島素敏感性正相關(Matsuda指數r=0.32),而DNMT3A則呈負相關(r=-0.28)。
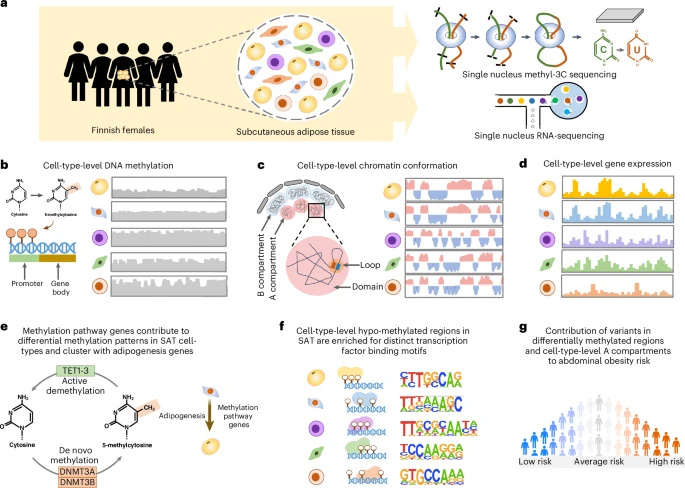
圖2 本研究采用 snm3C-seq 和 snRNA-seq 技術,在人類皮下脂肪組織(SAT)中對細胞類型水平的 DNA 甲基化、染色質構象及基因表達進行表征,并解析腹部肥胖的遺傳風險
肥胖風險的細胞類型特異性遺傳基礎
英國生物銀行(UKB)39萬人數據分析顯示,脂肪細胞低甲基化區域和A區室的變異分別解釋腰臀比調整BMI(WHRadjBMI)遺傳風險的82%和79%,顯著優于隨機背景(P<0.001)。髓系細胞a區室變異則與c反應蛋白(crp)水平顯著相關,印證了肥胖相關炎癥的髓系細胞起源假說。gwas變異富集分析發現,脂肪細胞dmrs中肥胖相關變異達3.2倍富集(p=6.5×10-8)。
這項研究首次在單細胞層面揭示SAT表觀遺傳異質性,建立DNA甲基化-三維基因組-轉錄組的調控網絡,為理解肥胖及其并發癥的細胞類型特異性機制提供了新視角。發現的過渡態細胞群體和TET1-DNMT3A調控軸,為靶向干預代謝疾病提供了潛在新靶點。
參考資料
[1] Single-cell DNA methylome and 3D genome atlas of human subcutaneous adipose tissue
摘要:研究通過snm3C-seq結合snRNA-seq,首次繪制了人類皮下脂肪組織(SAT)的細胞類型分辨率表觀基因組圖譜。
多組學解析人類皮下脂肪細胞異質性
研究團隊采用單核甲基化-3C測序(snm3C-seq)技術,對5例芬蘭女性皮下脂肪組織(SAT)活檢樣本進行多組學分析,同步捕獲6,652個核的DNA甲基化(mCG)和染色質構象數據,并與29,423個核的snRNA-seq數據整合。通過5kb分辨率mCG分析和100kb染色質互作聚類,鑒定出脂肪細胞、脂肪干細胞祖細胞(ASPCs)、血管周圍細胞等7種主要細胞類型,并發現63個核呈現表觀遺傳"過渡態"特征——其染色質構象類似脂肪細胞而甲基化模式接近血管周圍細胞,暗示人類脂肪細胞可能通過該路徑分化。

圖1 人類皮下脂肪組織的單細胞 DNA 甲基化組與三維基因組圖譜
甲基化與轉錄組的跨模態調控網絡
基因體mCG低甲基化與高表達呈現強相關性(如脂肪標記基因GPAM)。跨模態分析發現PPAR信號通路關鍵基因(ACSL1、ADIPOQ等)在脂肪細胞中同步呈現低mCG和高表達,而脂肪分化基因(如LEP、SREBF1)則富集于脂肪細胞特異性低甲基化區域。值得注意的是,僅21%的脂肪細胞標記基因在兩種模態中重疊,但63%相關生物學通路(如脂質代謝)具有跨模態一致性,提示表觀遺傳與轉錄調控的協同作用。
髓系與脂肪細胞的甲基化"蹺蹺板"效應
全基因組共鑒定705,063個差異甲基化區域(DMRs),覆蓋5.39%基因組。脂肪細胞與ASPCs分別呈現56.3%和50.6%區域低甲基化,而髓系細胞則73.0%區域高甲基化,47.3% DMRs呈現此消彼長模式。HOMER分析揭示髓系細胞低甲基化區域富集IRF4、MEF2B等轉錄因子結合基序,ENCODE的ChIP-seq數據驗證這些基序在GM12878細胞中富集度達3.5倍(P<1×10-12)。
脂肪細胞特異的基因組三維結構
脂肪細胞表現出獨特的染色質空間特征:短程互作(100kb-2Mb)比例較其他細胞高1.51倍(P<10-91),25kb分辨率下檢測到更多結構域(平均4,120個/細胞)。染色體區室分析顯示,脂肪細胞的A區室(活性染色質)與ADIPOQ等肥胖GWAS變異顯著共定位,而髓系細胞A區室則富集炎癥相關變異。值得注意的是,11,571個差異區室區域中,ADIPOQ基因所在基因組區域在ASPCs中處于B區室(抑制狀態),而在脂肪細胞中轉為A區室。
TET1-DNMT3A的表觀遺傳調控軸
單細胞轉錄組顯示去甲基化酶TET1在脂肪細胞特異性高表達(P<10-300),而甲基轉移酶DNMT3A在髓系細胞優勢表達(P<10-136)。人類前脂肪細胞分化實驗證實,TET1與PPARγ等脂肪生成基因呈現同步表達波動。SAT批量RNA-seq進一步發現,TET1表達與胰島素敏感性正相關(Matsuda指數r=0.32),而DNMT3A則呈負相關(r=-0.28)。

圖2 本研究采用 snm3C-seq 和 snRNA-seq 技術,在人類皮下脂肪組織(SAT)中對細胞類型水平的 DNA 甲基化、染色質構象及基因表達進行表征,并解析腹部肥胖的遺傳風險
肥胖風險的細胞類型特異性遺傳基礎
英國生物銀行(UKB)39萬人數據分析顯示,脂肪細胞低甲基化區域和A區室的變異分別解釋腰臀比調整BMI(WHRadjBMI)遺傳風險的82%和79%,顯著優于隨機背景(P<0.001)。髓系細胞a區室變異則與c反應蛋白(crp)水平顯著相關,印證了肥胖相關炎癥的髓系細胞起源假說。gwas變異富集分析發現,脂肪細胞dmrs中肥胖相關變異達3.2倍富集(p=6.5×10-8)。
這項研究首次在單細胞層面揭示SAT表觀遺傳異質性,建立DNA甲基化-三維基因組-轉錄組的調控網絡,為理解肥胖及其并發癥的細胞類型特異性機制提供了新視角。發現的過渡態細胞群體和TET1-DNMT3A調控軸,為靶向干預代謝疾病提供了潛在新靶點。
參考資料
[1] Single-cell DNA methylome and 3D genome atlas of human subcutaneous adipose tissue



