摘要:研究人員利用人工智能識別了古菌中此前未知的化合物。
它們在沸騰的酸液、深海噴口和鹽灘中生存了數十億年。如今,地球上一些最古老的生命形式——被稱為古菌的微生物——正在為對抗當今最緊迫的健康威脅之一——抗生素耐藥性——提供一種新的武器。
在《自然微生物學》雜志發表的一項新研究中,賓夕法尼亞大學的研究人員利用人工智能識別古菌中以前未知的化合物,這些化合物可以促進下一代抗生素的開發。
賓夕法尼亞大學工程與應用科學學院(Penn Engineering)生物工程和化學與生物分子工程系副教授、佩雷爾曼醫學院精神病學和微生物學系副教授、藝術與科學學院化學系副教授、該論文的資深作者César de la Fuente說道:“以前尋找新抗生素的努力主要關注真菌、細菌和動物。”
過去,de la Fuente 的實驗室曾利用人工智能模型從一系列意想不到的來源中識別候選抗生素,從滅絕生物的 DNA到動物毒液中的化學物質。現在,他們將這些工具應用于一組新的數據:數百種古代微生物的蛋白質。de la Fuente 說:“生命中還有另一個全新的領域等待探索。”

圖1 深度學習揭示古菌蛋白質組中的抗生素
探索微生物前沿
與細菌和真核生物(包括植物、動物和真菌)不同,古菌在生命之樹上占據著自己的分支。
盡管在顯微鏡下,古菌與細菌相似,但它們在基因、細胞膜和生物化學方面有著根本的不同。這些差異使它們能夠在地球上一些最極端的環境中生存,從過熱的海底熱泉到黃石國家公園那樣的酷熱溫泉。
由于古菌常常在其他生物難以生存的環境中茁壯成長——承受巨大的壓力、有毒化學物質和極端溫度——它們的生物學特性以不同尋常的方式進化。這使得它們成為一個充滿希望但尚未開發的新型分子工具來源,包括一些可能像抗生素一樣起作用但作用機制與目前使用的抗生素不同的化合物。
“我們之所以對古菌感興趣,是因為它們必須在特殊環境下進化出生化防御機制,”德拉富恩特實驗室的研究員、論文共同第一作者Marcelo Torres說道。“我們想,如果它們在那樣的條件下存活了數十億年,或許它們已經進化出了獨特的方法來對抗微生物競爭對手,或許我們可以從中學習。”
利用人工智能尋找抗生素
為了發現古菌中隱藏的潛在抗生素化合物,研究人員求助于人工智能。該團隊利用了APEX的更新版本,這是de la Fuente實驗室最初開發的一種人工智能工具,用于識別古生物學中的候選抗生素,包括猛犸象等已滅絕動物的蛋白質。
在觀察了數千種具有已知抗菌特性的肽(氨基酸短鏈)后,APEX 可以預測給定氨基酸序列產生類似效果的可能性。
通過對數千種額外的肽和有關導致人類疾病的細菌的信息對 APEX 1.1 進行重新訓練,研究人員準備了該工具來預測古細菌中哪些肽可能會抑制細菌的生長。
掃描233種古菌,發現了超過12000種候選抗生素。研究人員將這些分子命名為“古菌素”,化學分析顯示,它們與已知的抗菌肽(AMP)存在差異,尤其是在電荷分布方面。
研究人員隨后選擇了80種古菌素來對抗實際細菌。“試圖一次找到一個分子的新抗生素就像大海撈針,” de la Fuente實驗室的博士后研究員、該論文的另一位共同第一作者Fangping Wan說。“人工智能通過識別針可能的位置來加快這一過程。”
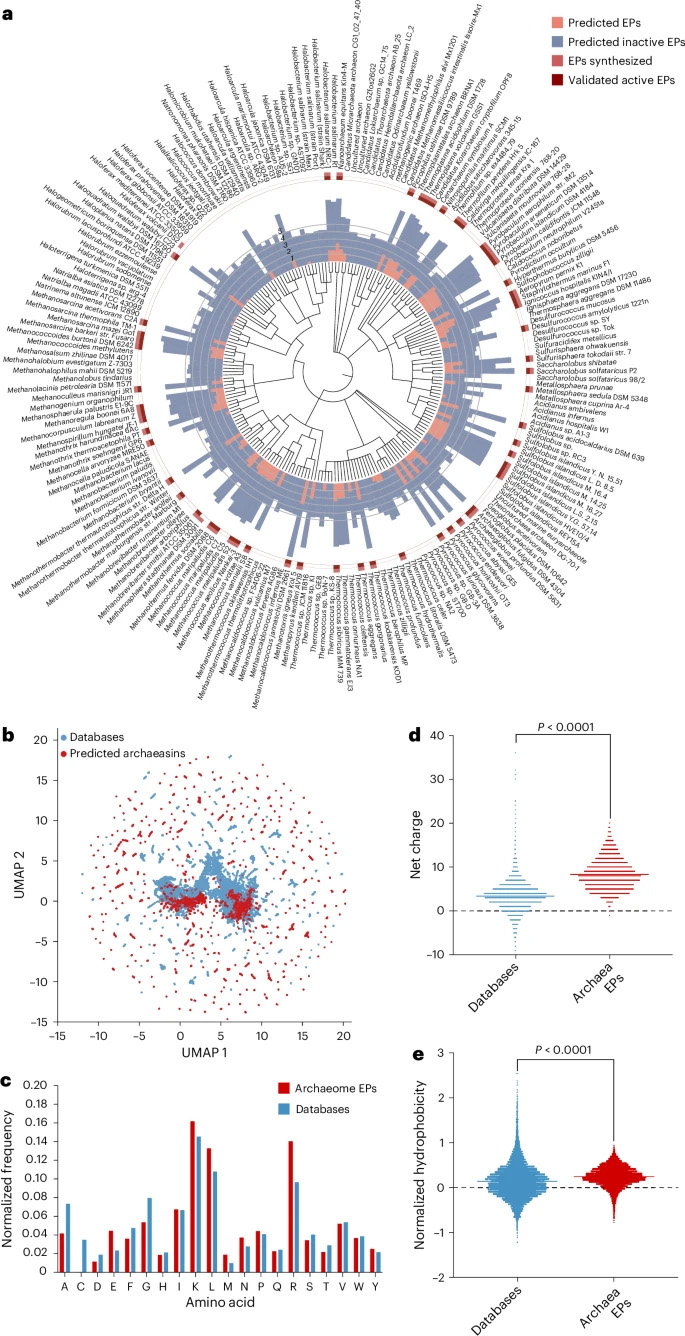
圖2 APEX人工智能模型對古菌組(古菌基因組/古菌群落組)的探索
與現有抗生素相當
抗生素的作用機制多種多樣。有些抗生素會在細菌細胞膜上打孔,而有些則會抑制細菌制造蛋白質的能力。研究人員發現,與大多數已知的攻擊細菌外部防御系統的AMP不同,古菌素似乎從細菌內部拔掉插頭,擾亂維持細胞存活的電信號。
在針對一系列致病耐藥細菌的測試中,80種古菌素中93%表現出對至少一種細菌的抗菌活性。研究人員隨后選擇了三種古菌素在動物模型中進行測試。
單次給藥四天后,這些古菌素均能抑制一種常在醫院內感染的耐藥細菌的傳播。其中一種化合物表現出與多粘菌素B相當的活性,多粘菌素B是一種常用于對抗耐藥性感染的最后一道防線的抗生素。
“這項研究表明,古菌中可能蘊藏著許多抗生素等待被發現,隨著越來越多的細菌對現有抗生素產生耐藥性,在非常規場所尋找新的抗生素來替代它們至關重要。”
未來研究
接下來,研究人員計劃進一步增強 APEX,使其能夠根據抗生素的結構預測候選藥物,從而提升該工具的準確性。研究人員還希望更好地了解古菌素的長期療效和安全性,并有朝一日將其應用于人體臨床試驗。
“這僅僅是個開始,古菌是最古老的生命形式之一,顯然有很多東西可以教我們如何戰勝當今面臨的病原體。”
參考資料
[1] Deep learning reveals antibiotics in the archaeal proteome
摘要:研究人員利用人工智能識別了古菌中此前未知的化合物。
它們在沸騰的酸液、深海噴口和鹽灘中生存了數十億年。如今,地球上一些最古老的生命形式——被稱為古菌的微生物——正在為對抗當今最緊迫的健康威脅之一——抗生素耐藥性——提供一種新的武器。
在《自然微生物學》雜志發表的一項新研究中,賓夕法尼亞大學的研究人員利用人工智能識別古菌中以前未知的化合物,這些化合物可以促進下一代抗生素的開發。
賓夕法尼亞大學工程與應用科學學院(Penn Engineering)生物工程和化學與生物分子工程系副教授、佩雷爾曼醫學院精神病學和微生物學系副教授、藝術與科學學院化學系副教授、該論文的資深作者César de la Fuente說道:“以前尋找新抗生素的努力主要關注真菌、細菌和動物。”
過去,de la Fuente 的實驗室曾利用人工智能模型從一系列意想不到的來源中識別候選抗生素,從滅絕生物的 DNA到動物毒液中的化學物質。現在,他們將這些工具應用于一組新的數據:數百種古代微生物的蛋白質。de la Fuente 說:“生命中還有另一個全新的領域等待探索。”

圖1 深度學習揭示古菌蛋白質組中的抗生素
探索微生物前沿
與細菌和真核生物(包括植物、動物和真菌)不同,古菌在生命之樹上占據著自己的分支。
盡管在顯微鏡下,古菌與細菌相似,但它們在基因、細胞膜和生物化學方面有著根本的不同。這些差異使它們能夠在地球上一些最極端的環境中生存,從過熱的海底熱泉到黃石國家公園那樣的酷熱溫泉。
由于古菌常常在其他生物難以生存的環境中茁壯成長——承受巨大的壓力、有毒化學物質和極端溫度——它們的生物學特性以不同尋常的方式進化。這使得它們成為一個充滿希望但尚未開發的新型分子工具來源,包括一些可能像抗生素一樣起作用但作用機制與目前使用的抗生素不同的化合物。
“我們之所以對古菌感興趣,是因為它們必須在特殊環境下進化出生化防御機制,”德拉富恩特實驗室的研究員、論文共同第一作者Marcelo Torres說道。“我們想,如果它們在那樣的條件下存活了數十億年,或許它們已經進化出了獨特的方法來對抗微生物競爭對手,或許我們可以從中學習。”
利用人工智能尋找抗生素
為了發現古菌中隱藏的潛在抗生素化合物,研究人員求助于人工智能。該團隊利用了APEX的更新版本,這是de la Fuente實驗室最初開發的一種人工智能工具,用于識別古生物學中的候選抗生素,包括猛犸象等已滅絕動物的蛋白質。
在觀察了數千種具有已知抗菌特性的肽(氨基酸短鏈)后,APEX 可以預測給定氨基酸序列產生類似效果的可能性。
通過對數千種額外的肽和有關導致人類疾病的細菌的信息對 APEX 1.1 進行重新訓練,研究人員準備了該工具來預測古細菌中哪些肽可能會抑制細菌的生長。
掃描233種古菌,發現了超過12000種候選抗生素。研究人員將這些分子命名為“古菌素”,化學分析顯示,它們與已知的抗菌肽(AMP)存在差異,尤其是在電荷分布方面。
研究人員隨后選擇了80種古菌素來對抗實際細菌。“試圖一次找到一個分子的新抗生素就像大海撈針,” de la Fuente實驗室的博士后研究員、該論文的另一位共同第一作者Fangping Wan說。“人工智能通過識別針可能的位置來加快這一過程。”

圖2 APEX人工智能模型對古菌組(古菌基因組/古菌群落組)的探索
與現有抗生素相當
抗生素的作用機制多種多樣。有些抗生素會在細菌細胞膜上打孔,而有些則會抑制細菌制造蛋白質的能力。研究人員發現,與大多數已知的攻擊細菌外部防御系統的AMP不同,古菌素似乎從細菌內部拔掉插頭,擾亂維持細胞存活的電信號。
在針對一系列致病耐藥細菌的測試中,80種古菌素中93%表現出對至少一種細菌的抗菌活性。研究人員隨后選擇了三種古菌素在動物模型中進行測試。
單次給藥四天后,這些古菌素均能抑制一種常在醫院內感染的耐藥細菌的傳播。其中一種化合物表現出與多粘菌素B相當的活性,多粘菌素B是一種常用于對抗耐藥性感染的最后一道防線的抗生素。
“這項研究表明,古菌中可能蘊藏著許多抗生素等待被發現,隨著越來越多的細菌對現有抗生素產生耐藥性,在非常規場所尋找新的抗生素來替代它們至關重要。”
未來研究
接下來,研究人員計劃進一步增強 APEX,使其能夠根據抗生素的結構預測候選藥物,從而提升該工具的準確性。研究人員還希望更好地了解古菌素的長期療效和安全性,并有朝一日將其應用于人體臨床試驗。
“這僅僅是個開始,古菌是最古老的生命形式之一,顯然有很多東西可以教我們如何戰勝當今面臨的病原體。”
參考資料
[1] Deep learning reveals antibiotics in the archaeal proteome



